多模态数据的整合计算是数据科学和机器学习研究的热点问题。其中,整合单细胞多模态测序数据, 进行全面挖掘和解析,是单细胞数据分析的基础和核心环节之一。由于不同模态数据集间样本(细胞)缺少匹配信息,以及数据集间存在异质性,使得数据整合非常具有挑战性。
2022年12月1日,中国科学院数学与系统科学研究院万林课题组联合来自上海交通大学以及同济大学的研究人员,在《自然·通讯》(Nature Communications)期刊发表了题为“一种基于最优传输整合单细胞数据的统一计算框架”(A unified computational framework for single-cell data integration with optimal transport)的研究论文。该论文基于耦合变分自编器(VAE)的深度学习技术和小批量非平衡最优传输(Minibatch-UOT)技术,提出了一种准确、高效、适用于整合大规模多模态异质数据的统一计算框架uniPort。
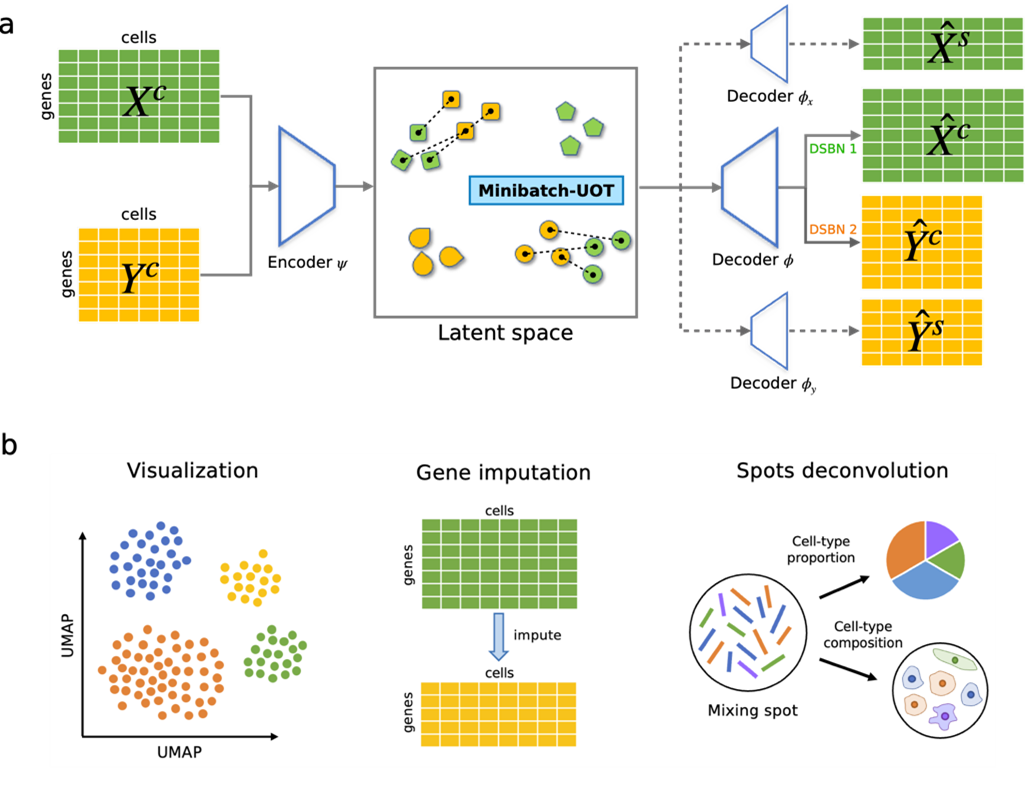
具体而言,uniPort有以下几点优势:(1) 利用多模态数据集的公共高度可变基因以及各个数据集的特有高度可变基因进行系统建模,从而在模型训练过程中能够对异质细胞群体在隐空间中进行高精度的鉴定与分离; (2) 采用了新型耦合 VAE 神经网络模型,增强了方法的非线性校正能力,并利用耦合 VAE 的泛化能力构建了跨模态在线预测的参考图谱; (3) 最小化 Minibatch-UOT 损失,一方面满足了大规模数据的计算需求,一方面实现了对异质数据集的内嵌结构对齐,解决了其他基于自编码器的多模态数据整合方法依赖样本匹配先验信息的局限; (4) 输出全局最优传输的概率转移矩阵,即细胞-细胞匹配矩阵,为空间异质数据的反卷积提供了灵活与有效的迁移学习基础。
该方法整合了多个单细胞多模态数据集和空间转录组数据集,解析小鼠大脑皮层神经元的空间结构,以及肿瘤的空间结构及其微环境等,取得了较好的应用。
综上所述,该工作开发了单细胞多模态数据整合统一的计算框架uniPort,可以将不同模态的单细胞测序数据映射到统一的细胞嵌入空间,并利用最优传输技术有效处理生物学差异和模态间的异质性。该方法将对生物医学中单细胞测序数据的精确解析提供基础支持。
论文信息:Cao, K., Gong, Q., Hong, Y., Wan, L. A unified computational framework for single-cell data integration with optimal transport. Nature Communications, 13, 7419 (2022).
论文链接:https://doi.org/10.1038/s41467-022-35094-8
附件下载: